Heat Wave and Cold Spells
Dieses Skript verarbeitet den Datensatz „Heat Wave Days“ aus dem Copernics Climate Data Store. Der Datensatz enthält die Anzahl der Hitzewellen-Tage (Heat wave days; HWD), die mit verschiedenen europaweiten sowie nationalen/regionalen Definitionen im Rahmen des C3S European Health Service entwickelt wurden. Diese Tage sind für unterschiedliche zukünftige Zeiträume und Klimawandelszenarien verfügbar.
Informationen zum Datensatz:
Source: Heat Wave and Cold Spells
Author: T. Tewes (Stadt Konstanz)
Notebook-Version: 1.2 (Aktualisiert: März 05, 2024)
1. Festlegen der Pfade und Arbeitsverzeichnisse
import os
''' ---- Verzeichnisse hier angeben ---- '''
download_folder = r".\data\sis-heat-and-cold-spells\download"
working_folder = r".\data\sis-heat-and-cold-spells\working"
geotiff_folder = r".\data\sis-heat-and-cold-spells\geotiff"
csv_folder = r".\data\sis-heat-and-cold-spells\csv"
output_folder = r".\data\sis-heat-and-cold-spells\output"
''' ----- Ende der Angaben ---- '''
os.makedirs(download_folder, exist_ok=True)
os.makedirs(working_folder, exist_ok=True)
os.makedirs(geotiff_folder, exist_ok=True)
os.makedirs(csv_folder, exist_ok=True)
os.makedirs(output_folder, exist_ok=True)
2. Herunterladen und Entpacken des Datensatzes
2.1 Authentifizierung
import cdsapi
def main():
# API-Key für die Authentifizierung
api_key = "fdae60fd-35d4-436f-825c-c63fedab94a4"
api_url = "https://cds.climate.copernicus.eu/api"
# Erstellung des CDS-API-Clients
client = cdsapi.Client(url=api_url, key=api_key)
return client
2.2 Definieren Sie die „request“ und laden Sie den Datensatz herunter
# Definieren der Begrenzungsrahmen-Koordinaten (WGS84-Format)
# Das Koordinatenformat lautet: [Norden, Westen, Süden, Osten]
bbox_wgs84_deutschland = [56.0, 5.8, 47.2, 15.0]
bbox_wgs84_de_standard = [5.7, 47.1, 15.2, 55.2]
bbox_wgs84_konstanz = [47.9, 8.9, 47.6, 9.3]
bbox_wgs84_konstanz_standard = [9.0, 47.6, 9.3, 47.8] # [West, South, East, North]
# Alternativ können Sie ein Shapefile für eine präzise geografische Filterung verwenden
import geopandas as gpd
import math
# Beispiel: Shapefile von Konstanz laden (WGS84-Projektion)
de_shapefile = r"./shapefiles/de_boundary.shp"
de_gdf = gpd.read_file(de_shapefile)
# Extrahieren Sie den Begrenzungsrahmen des Shapefiles
de_bounds = de_gdf.total_bounds
# Passen Sie den Begrenzungsrahmen an und puffern Sie ihn, um einen etwas größeren
de_bounds_adjusted = [(math.floor(de_bounds[0]* 10)/10)-0.1,
(math.floor(de_bounds[1]* 10)/10)-0.1,
(math.ceil(de_bounds[2]* 10)/10)+0.1,
(math.ceil(de_bounds[3]* 10)/10)+0.1]
# Ordnen Sie die Koordinaten in das Format: [Nord, West, Süd, Ost] um.
bbox_de_bounds_adjusted = [de_bounds_adjusted[3], de_bounds_adjusted[0],
de_bounds_adjusted[1], de_bounds_adjusted[2]]
# Der Datensatz sis-heat-and-cold-spells ermöglicht die Auswahl zwischen zwei Variablen:
# "Heat wave days" and "Cold spell days".
# Abhängig von der gewählten Variablen variieren die Definitionsoptionen.
# Für "heat_wave_days" sind alle Definitionen verfügbar.
# Für "cold_spell_days" wird nur die 'landesspezifische' Definition unterstützt.
variable_to_definition_map = {
'heat_wave_days': ['climatological_related', 'health_related', 'country_related'],
'cold_spell_days': ['country_related']
}
# Ausgewählte Variable
selected_variable = 'heat_wave_days'
# Abrufen der entsprechenden Definitionen basierend auf der ausgewählten Variablen
selected_definition = variable_to_definition_map.get(selected_variable, [])
# Ausgabe der ausgewählten Definitionen und der Variable
print(f"ausgewählte_variable: {selected_variable}\nausgewählte_definition: {selected_definition}")
ausgewählte_variable: heat_wave_days
ausgewählte_definition: ['climatological_related', 'health_related', 'country_related']
# Definition des Datensatzes und der Request-Parameter
dataset = "sis-heat-and-cold-spells"
request = {
"variable": selected_variable,
"definition": selected_definition,
"experiment": [
"rcp4_5",
"rcp8_5"
],
"ensemble_statistic": [
"ensemble_members_average",
"ensemble_members_standard_deviation"
],
"area": bbox_de_bounds_adjusted
}
# Führen Sie es aus, um den Datensatz herunterzuladen:
def main_retrieve():
dataset_filename = f"{dataset}-{selected_definition}.zip"
dataset_filepath = os.path.join(download_folder, dataset_filename)
# Den Datensatz nur herunterladen, wenn er noch nicht heruntergeladen wurde
if not os.path.isfile(dataset_filepath):
# Rufen Sie den CDS-Client nur auf, wenn der Datensatz noch nicht heruntergeladen wurde.
client = main()
# Den Datensatz mit den definierten Anforderungsparametern herunterladen
client.retrieve(dataset, request, dataset_filepath)
else:
print("Datensatz bereits heruntergeladen.")
if __name__ == "__main__":
main_retrieve()
Datensatz bereits heruntergeladen.
2.3 Extrahieren die ZIP-Datei in Ordner
import zipfile
# Definieren Sie einen Extraktionsordner für die ZIP-Datei, der dem Arbeitsordner entspricht
extract_folder = os.path.join(working_folder, f"{selected_variable}")
os.makedirs(extract_folder, exist_ok=True)
# Extract the zip file
try:
if not os.listdir(extract_folder):
dataset_filename = f"{dataset}-{selected_definition}.zip"
dataset_filepath = os.path.join(download_folder, dataset_filename)
with zipfile.ZipFile(dataset_filepath, 'r') as zip_ref:
zip_ref.extractall(extract_folder)
print(f"Dateien erfolgreich extrahiert nach: {extract_folder}")
else:
print("Ordner ist nicht leer. Entpacken überspringen.")
except FileNotFoundError:
print(f"Fehler: Die Datei {dataset_filepath} wurde nicht gefunden.")
except zipfile.BadZipFile:
print(f"Fehler: Die Datei {dataset_filepath} ist keine gültige ZIP-Datei.")
except Exception as e:
print(f"Ein unerwarteter Fehler ist aufgetreten: {e}")
Ordner ist nicht leer. Entpacken überspringen.
3. Untersuchen der Metadaten der NetCDF4-Datei
⚠️ Wichtig: Obwohl alle Datensätze zum Download verfügbar sind, konzentriert sich dieses Notebook auf die Analyse der folgenden Auswahl:
selected_variable = ‚heat_wave_days‘
selected_definition = ‚climatological related‘
# Variable und Definition definieren
selected_variable = 'heat_wave_days'
selected_definition = 'climatological_related'
3.1 Erstellen eines DataFrame mit verfügbaren NetCDF-Dateien
import re
import pandas as pd
import netCDF4 as nc
def meta(filename):
# Überprüfen, ob der Dateiname dem erwarteten Muster entspricht
match = re.search(r"(?P<ds_variable>\w+?)_(?P<ds_definition>\w+?)_(?P<rcp>rcp\d+?)_(?P<rcp_statistic>mean|stdev)_v(\d+\.\d+)\.", filename)
# Fehler ausgeben, wenn der Dateiname nicht dem erwarteten Schema entspricht
if not match:
match = re.search("Der angegebene Dateiname entspricht nicht dem erwarteten Benennungsschema.")
# Funktion zum Extrahieren des Variablennamens aus der NetCDF-Datei
def get_nc_variable():
with nc.Dataset(os.path.join(extract_folder, filename), 'r') as nc_dataset:
nc_variable_name = nc_dataset.variables.keys()
return [*nc_variable_name][0]
# Metadaten als Dictionary zurückgeben
return dict(
filename=filename,
path=os.path.join(extract_folder, filename),
ds_variable=match.group('ds_variable'),
ds_definition=match.group('ds_definition'),
variable_name=get_nc_variable(),
rcp=match.group('rcp'),
rcp_statistic=match.group('rcp_statistic')
)
# Metadaten für alle NetCDF-Dateien im Ordner extrahieren
# Das Dictionary 'nc_files' enthält alle relevanten Metadaten der verfügbaren NetCDF4-Dateien
# Dieses Dictionary wird später verwendet, um die Dateien in GeoTiff zu konvertieren
nc_files = [meta(f) for f in os.listdir(extract_folder) if f.endswith('.nc')]
df_nc_files = pd.DataFrame.from_dict(nc_files)
# Pandas-Anzeigeoptionen anpassen
pd.options.display.max_colwidth = 24
# DataFrame anzeigen, ohne die Spalte 'path' darzustellen
df_nc_files.loc[:, df_nc_files.columns != 'path']
| filename | ds_variable | ds_definition | variable_name | rcp | rcp_statistic | |
|---|---|---|---|---|---|---|
| 0 | HWD_EU_climate_rcp45... | HWD | EU_climate | HWD_EU_climate | rcp45 | mean |
| 1 | HWD_EU_climate_rcp45... | HWD | EU_climate | HWD_EU_climate | rcp45 | stdev |
| 2 | HWD_EU_climate_rcp85... | HWD | EU_climate | HWD_EU_climate | rcp85 | mean |
| 3 | HWD_EU_climate_rcp85... | HWD | EU_climate | HWD_EU_climate | rcp85 | stdev |
| 4 | HWD_EU_health_rcp45_... | HWD | EU_health | HWD_EU_health | rcp45 | mean |
| 5 | HWD_EU_health_rcp45_... | HWD | EU_health | HWD_EU_health | rcp45 | stdev |
| 6 | HWD_EU_health_rcp85_... | HWD | EU_health | HWD_EU_health | rcp85 | mean |
| 7 | HWD_EU_health_rcp85_... | HWD | EU_health | HWD_EU_health | rcp85 | stdev |
| 8 | HWD_national_rcp45_m... | HWD | national | HWD_merged | rcp45 | mean |
| 9 | HWD_national_rcp45_s... | HWD | national | HWD_merged | rcp45 | stdev |
| 10 | HWD_national_rcp85_m... | HWD | national | HWD_merged | rcp85 | mean |
| 11 | HWD_national_rcp85_s... | HWD | national | HWD_merged | rcp85 | stdev |
3.2 Einzigartige Variablennamen und verfügbare Variablen ausgeben
# Variable definieren, um bereits verarbeitete Variablennamen zu speichern und Duplikate zu vermeiden
seen_variables = set()
# Alle Variablen in jeder NetCDF-Datei auflisten
for i, nc_file in enumerate(nc_files):
variable_name = nc_file['variable_name']
# Überspringen, wenn die Variable bereits verarbeitet wurde
if variable_name in seen_variables:
continue
# NetCDF-Datei im Lesemodus öffnen
with nc.Dataset(nc_file['path'], mode='r') as nc_dataset:
# Alle Variablen im aktuellen Datensatz auflisten
variables_list = list(nc_dataset.variables.keys())
# Details der Datei und ihrer Variablen ausgeben
print(f"{i + 1:<2} {variable_name:<18}: Verfügbare Variablen: {variables_list}")
# Diese Variable als verarbeitet markieren
seen_variables.add(variable_name)
1 HWD_EU_climate : Verfügbare Variablen: ['HWD_EU_climate', 'height', 'quantile', 'lat', 'lon', 'time']
5 HWD_EU_health : Verfügbare Variablen: ['HWD_EU_health', 'height', 'lat', 'lon', 'time']
9 HWD_merged : Verfügbare Variablen: ['HWD_merged', 'height', 'lat', 'lon', 'time']
# Alle Variableninformationen in jeder NetCDF-Datei auflisten
seen_variables = set()
# Alle variablen Informationen in jeder NetCDF-Datei auflisten
for i, nc_file in enumerate(nc_files):
variable_name = nc_file['variable_name']
# Überspringen, wenn die Variable bereits verarbeitet wurde
if variable_name in seen_variables:
continue
# NetCDF-Datei im Lesemodus öffnen
with nc.Dataset(nc_file['path'], mode='r') as nc_dataset:
# Primärvariable-Daten abrufen
variable_data = nc_dataset[variable_name]
# Zusammenfassung der Primärvariable erstellen
summary = {
"Variablenname": variable_name,
"Datentyp": variable_data.dtype,
"Form": variable_data.shape,
"Variableninfo": f"{variable_data.dimensions}",
"Einheiten": getattr(variable_data, "units", "N/A"),
"Langer Name": getattr(variable_data, "long_name", "N/A"),
}
# Datensatz-Zusammenfassung als DataFrame zur besseren Visualisierung anzeigen
nc_summary = pd.DataFrame(list(summary.items()), columns=['Beschreibung', 'Bemerkungen'])
print(f"{i + 1}. Zusammenfassung der Variable '{variable_name}':")
display(nc_summary)
# Variablenname zur Liste der bereits verarbeiteten Variablen hinzufügen
seen_variables.add(variable_name)
# Ausgabe begrenzen
output_limit = 2
if len(seen_variables) >= output_limit:
print(f".... (Ausgabe auf die ersten {output_limit} Variablen gekürzt)")
break
1. Zusammenfassung der Variable 'HWD_EU_climate':
| Beschreibung | Bemerkungen | |
|---|---|---|
| 0 | Variablenname | HWD_EU_climate |
| 1 | Datentyp | float32 |
| 2 | Form | (100, 82, 95) |
| 3 | Variableninfo | ('time', 'lat', 'lon') |
| 4 | Einheiten | day |
| 5 | Langer Name | Ensemble members ave... |
5. Zusammenfassung der Variable 'HWD_EU_health':
| Beschreibung | Bemerkungen | |
|---|---|---|
| 0 | Variablenname | HWD_EU_health |
| 1 | Datentyp | float32 |
| 2 | Form | (100, 82, 95) |
| 3 | Variableninfo | ('time', 'lat', 'lon') |
| 4 | Einheiten | day |
| 5 | Langer Name | Ensemble members ave... |
.... (Ausgabe auf die ersten 2 Variablen gekürzt)
4. Exportieren der NetCDF4-Dateien im CSV-Format
4.1 Definieren eine Funktion zum Konvertieren von NetCDF-Daten in DataFrame
import xarray as xr
# Funktion zur Konvertierung von NetCDF-Daten in ein Pandas DataFrame
def netcdf_to_dataframe(
nc_file,
bounding_box=None):
# Öffne das NetCDF-Dataset im Lesemodus
with xr.open_dataset(nc_file['path']) as nc_dataset:
# Zugriff auf die Variablendaten aus dem Datensatz
variable_data = nc_dataset[nc_file['variable_name']]
# Sicherstellen, dass die Namen für Breiten- und Längengrad korrekt sind
latitude_name = 'latitude' if 'latitude' in nc_dataset.coords else 'lat'
longitude_name = 'longitude' if 'longitude' in nc_dataset.coords else 'lon'
# Falls eine Begrenzungsbox angegeben ist, die Daten filtern
if bounding_box:
filtered_data = variable_data.where(
(nc_dataset[latitude_name] >= bounding_box[1]) & (nc_dataset[latitude_name] <= bounding_box[3]) &
(nc_dataset[longitude_name] >= bounding_box[0]) & (nc_dataset[longitude_name] <= bounding_box[2]),
drop=True
)
else:
filtered_data = variable_data
# Umwandlung des xarray-Datensatzes in ein Pandas DataFrame
df = filtered_data.to_dataframe().reset_index().set_index(['time', latitude_name, longitude_name])
variable_column_name = f"{nc_file['variable_name']}_{nc_file['rcp']}_{nc_file['rcp_statistic']}"
df.rename(columns={nc_file['variable_name']: variable_column_name}, inplace=True)
# Entfernen nicht benötigter Spalten (variiert je nach Datensatz)
if 'height' in df.columns:
df = df.drop(columns=['height'])
if 'quantile' in df.columns:
df = df.drop(columns=['quantile'])
return df
4.2 DataFrame erstellen und als zusammengeführte CSV-Datei exportieren
from tqdm.notebook import tqdm
import textwrap
# Definieren Sie den Variablennamen für die Filterung von NetCDF-Dateien.
variable_name = 'HWD_EU_climate' # Andere Optionen: HWD_EU_climate, HWD_EU_health, HWD_merged (für HWD_national), oder CDS_national
# Erstellen Sie einen Ordner für die Speicherung von CSV-Teilsatzdateien.
subset_csv_folder = os.path.join(csv_folder, f"{variable_name}")
os.makedirs(subset_csv_folder, exist_ok=True) # Ensure the folder exists
# Definieren Sie den Namen der CSV-Ausgabedatei anhand des Variablennamens und der ausgewählten Definition
csv_filename = f"{variable_name}_{selected_definition}.csv"
csv_filepath = os.path.join(subset_csv_folder, csv_filename)
# Filtern Sie die Liste der NetCDF-Dateien so, dass nur diejenigen enthalten sind, die dem Variablennamen entsprechen
nc_files_subset = list(filter(lambda nc_file: nc_file['variable_name'] == variable_name, nc_files))
# Exportieren Sie die NetCDF-Dateien als zusammengeführte CSV-Datei
if not os.path.isfile(csv_filepath):
dataframes = [netcdf_to_dataframe(nc_file) for nc_file in tqdm(nc_files_subset)]
df_merged = pd.concat(dataframes, axis=1)
df_merged.to_csv(csv_filepath, sep=',', encoding='utf8')
else:
print(f"Datei existiert bereits unter {csv_filepath}.\nÜberspringen den Export.")
print("Lesen bestehende CSV-Datei ein...")
df_merged = pd.read_csv(csv_filepath).set_index(['time', 'lat', 'lon'])
# Funktion zum Umbruch der Spaltennamen für bessere Lesbarkeit
def wrap_column_names(df, width):
wrapped_columns = {col: " ".join(textwrap.wrap(col, width)) for col in df.columns}
return df.rename(columns=wrapped_columns)
# Ändere die Pandas-Anzeigeoptionen
pd.options.display.float_format = '{:,.2f}'.format
# Zeige das DataFrame an
df_wrapped = wrap_column_names(df_merged, width=14)
df_wrapped
Datei existiert bereits unter .\data\sis-heat-and-cold-spells\csv\HWD_EU_climate\HWD_EU_climate_climatological_related.csv.
Überspringen den Export.
Lesen bestehende CSV-Datei ein...
| HWD_EU_climate _rcp45_mean | HWD_EU_climate _rcp45_stdev | HWD_EU_climate _rcp85_mean | HWD_EU_climate _rcp85_stdev | |||
|---|---|---|---|---|---|---|
| time | lat | lon | ||||
| 1986-01-01 | 47.10 | 5.70 | 0.77 | 1.33 | 0.77 | 0.95 |
| 5.80 | 0.76 | 1.31 | 0.76 | 0.96 | ||
| 5.90 | 0.77 | 1.18 | 0.77 | 0.89 | ||
| 6.00 | 0.74 | 0.96 | 0.74 | 0.72 | ||
| 6.10 | 0.72 | 0.80 | 0.72 | 0.63 | ||
| ... | ... | ... | ... | ... | ... | ... |
| 2085-01-01 | 55.20 | 14.70 | 3.60 | 2.06 | 6.99 | 2.59 |
| 14.80 | 2.84 | 1.80 | 5.61 | 2.45 | ||
| 14.90 | 2.58 | 1.48 | 5.11 | 2.09 | ||
| 15.00 | 3.00 | 1.58 | 5.66 | 1.98 | ||
| 15.10 | 3.43 | 2.30 | 6.42 | 2.68 |
779000 rows × 4 columns
5. Exportieren der NetCDF4-Datei nach GeoTIFF
5.1 Definieren eine Funktion zum Exportieren der NetCDF4-Datei als GeoTIFF-Datei(en)
import numpy as np
from rasterio.transform import from_origin
import rasterio
from tqdm.notebook import tqdm
def main_export_geotiff(
nc_file,
bounding_box=None,
start_year=None,
end_year=None,
merged=None,
output_directory=None):
"""
Parameter:
nc_file (dict): Ein Wörterbuch mit den Schlüsseln 'path' (Dateipfad), 'variable', 'rcp' und 'statistic'.
bounding_box (list): [lon_min, lat_min, lon_max, lat_max] (optional).
start_year (int): Startjahr für den Datensatz (optional).
end_year (int): Endjahr für den Datensatz (optional).
merged (bool): Ob ein zusammengeführtes GeoTIFF oder einzelne GeoTIFFs erstellt werden sollen (optional).
output_directory (str): Verzeichnis zum Speichern der Ausgabe-GeoTIFF-Dateien (optional).
"""
# Öffnet die NetCDF-Datei
with nc.Dataset(nc_file['path'], 'r') as nc_dataset:
nc_dataset = nc.Dataset(nc_file['path'], 'r')
lon = nc_dataset['lon'][:]
lat = nc_dataset['lat'][:]
# Falls eine Begrenzungsbox angegeben wurde, filtere die Daten entsprechend
if bounding_box:
lon_min, lat_min, lon_max, lat_max = bounding_box
indices_lat = np.where((lat >= lat_min) & (lat <= lat_max))[0]
indices_lon = np.where((lon >= lon_min) & (lon <= lon_max))[0]
start_lat, end_lat = indices_lat[0], indices_lat[-1] + 1
start_lon, end_lon = indices_lon[0], indices_lon[-1] + 1
else:
start_lat, end_lat = 0, len(lat)
start_lon, end_lon = 0, len(lon)
lat = lat[start_lat:end_lat]
lon = lon[start_lon:end_lon]
# Extrahiere die Zeitvariable und konvertiere sie in lesbare Datumsangaben
time_var = nc_dataset.variables['time']
time_units = time_var.units
time_calendar = getattr(time_var, "calendar", "standard")
cftime = nc.num2date(time_var[:], units=time_units, calendar=time_calendar)
# Berechnet die räumliche Auflösung und die Rastertransformation
dx = abs(lon[1] - lon[0])
dy = abs(lat[1] - lat[0])
transform = from_origin(lon.min() - dx / 2, lat.min() - dy / 2, dx, -dy)
# Bestimmt den verfügbaren Zeitraum
min_year = cftime[0].year
max_year = cftime[-1].year
if start_year and end_year:
# Passen Sie start_year und end_year basierend auf dem verfügbaren Zeitraum an
if start_year < min_year:
print(f"Das angegebene Startjahr {start_year} liegt vor dem Datensatzbereich.")
print(f"Das Startjahr wird auf {min_year} angepasst.")
if end_year > max_year:
print(f"Das angegebene Endjahr {end_year} liegt nach dem Datensatzbereich.")
print(f"Das Endjahr wird auf {max_year} angepasst.")
start_year = max(start_year, min_year)
end_year = min(end_year, max_year)
else:
# Standardmäßig wird der gesamte Datensatz verwendet
start_year = min_year
end_year = max_year
# Findet die Indizes, die dem angegebenen Jahresbereich entsprechen
start_index = next(i for i, dt in enumerate(cftime) if dt.year == start_year)
end_index = next(i for i, dt in enumerate(cftime) if dt.year == end_year) + 1 # Full year (monthly data)
# Extrahiere Variablen-Daten
variable_data = nc_dataset.variables[nc_file['variable_name']]
variable_data_subset = variable_data[start_index:end_index, start_lat:end_lat, start_lon:end_lon]
if merged:
# Erstellt ein zusammengeführtes GeoTIFF mit allen Zeitscheiben als separate Bänder
if output_directory:
subset_directory_path = output_directory
else:
subset_directory_path = os.path.join(geotiff_folder, f"{selected_variable}_{selected_definition}-merged")
os.makedirs(subset_directory_path, exist_ok=True)
# Pfad der Ausgabedatei festlegen
output_filename = f"{nc_file['variable_name']}_{nc_file['rcp']}_{nc_file['rcp_statistic']}_{start_year}-{end_year}.tif"
output_filepath = os.path.join(subset_directory_path, output_filename)
# Erstellt eine GeoTIFF-Datei mit mehreren Bändern für jede Zeitscheibe
with rasterio.open(
output_filepath,
"w",
driver = "GTiff",
dtype = str(variable_data_subset.dtype),
width = variable_data_subset.shape[2],
height = variable_data_subset.shape[1],
count = variable_data_subset.shape[0],
crs = "EPSG:4326",
nodata = -9999,
transform=transform,
) as dst:
for year_index in tqdm(range(variable_data_subset.shape[0]),
desc=f"Exportiere zusammengeführte GeoTIFF-Datei von {start_year} bis {end_year}"):
band_data = variable_data_subset[year_index,:,:]
dt = cftime[start_index + year_index]
band_desc = f"{dt.year:04d}-{dt.month:02d}-{dt.day:02d}"
# Schreibe jede Jahresscheibe als Band
dst.write(band_data, year_index + 1)
dst.set_band_description(year_index + 1, band_desc)
else:
# Export als einzelne GeoTIFF-Dateien
if output_directory:
subset_directory_path = output_directory
else:
subset_directory_path = os.path.join(geotiff_folder, f"{selected_variable}_{selected_definition}-individual")
os.makedirs(subset_directory_path, exist_ok=True)
for year_index in tqdm(range(variable_data_subset.shape[0]),
desc="Exportieren einzelner GeoTIFF-Dateien"):
# Bestimmt das Datum für die aktuelle Zeitscheibe
dt = cftime[start_index + year_index]
dt_full = f"{dt.year:04d}-{dt.month:02d}-{dt.day:02d}"
# Definiert den Speicherort der Ausgabe-GeoTIFF-Datei
output_filename = f"{nc_file['variable_name']}_{nc_file['rcp']}_{nc_file['rcp_statistic']}_{dt_full}.tif"
output_filepath = os.path.join(subset_directory_path, output_filename)
# Exportiert die aktuelle Zeitscheibe als GeoTIFF
with rasterio.open(
output_filepath,
"w",
driver="GTiff",
dtype=str(variable_data_subset.dtype),
width=variable_data_subset.shape[2],
height=variable_data_subset.shape[1],
count=1,
crs="EPSG:4326",
nodata=-9999,
transform=transform,
) as dst:
year_precipitation_data = variable_data_subset[year_index, :, :]
dst.write(year_precipitation_data, 1)
dst.set_band_description(1, f"{dt.year:04d}-{dt.month:02d}-{dt.day:02d}")
5.2 Ausgewählte NetCDF4-Datei(en) in GeoTIFF-Datei(en) exportieren
if __name__ == "__main__":
# Exportieren Sie alle NetCDF-Dateien als zusammengeführte GeoTIFF-Dateien.
for nc_file in nc_files_subset:
main_export_geotiff(
nc_file=nc_file,
bounding_box=None,
merged=True
)
# Exportieren Sie alle NetCDF-Dateien als einzelne GeoTIFF-Dateien.
for nc_file in nc_files_subset:
main_export_geotiff(
nc_file=nc_file,
bounding_box=None,
start_year = 1995,
end_year = 2000,
merged = False,
)
# Zusätzlicher Fall (Erweiterte Filterung)
# temp_folder = os.path.join(geotiff_folder, "_temp_folder")
# os.makedirs(temp_folder, exist_ok=True)
# main_export_geotiff(
# nc_file=nc_files[0],
# bounding_box=bbox_wgs84_konstanz_standard,
# start_year=1995,
# end_year=2000,
# merged=True,
# output_directory=temp_folder)
6. Analyse und Visualisierung Optionen
6.1 Vorbereitung der Daten für die Visualisierung
# Daten für die Region Konstanz filtern (WGS84-Format)
lon_min, lat_min, lon_max, lat_max = bbox_wgs84_konstanz_standard
# DataFrame mit einer Abfrage filtern
filtered_df = (
df_merged.query(
"@lat_min <= lat <= @lat_max and @lon_min <= lon <= @lon_max"
)
.reset_index()
.set_index("time")
)
# DataFrame anzeigen
df_wrapped = wrap_column_names(filtered_df, width=11)
df_wrapped.head()
| lat | lon | HWD_EU_clim ate_rcp45_m ean | HWD_EU_clim ate_rcp45_s tdev | HWD_EU_clim ate_rcp85_m ean | HWD_EU_clim ate_rcp85_s tdev | |
|---|---|---|---|---|---|---|
| time | ||||||
| 1986-01-01 | 47.60 | 9.00 | 0.69 | 1.18 | 0.69 | 0.74 |
| 1986-01-01 | 47.60 | 9.10 | 0.68 | 1.17 | 0.68 | 0.76 |
| 1986-01-01 | 47.60 | 9.20 | 0.66 | 1.13 | 0.66 | 0.74 |
| 1986-01-01 | 47.70 | 9.00 | 0.67 | 1.16 | 0.67 | 0.71 |
| 1986-01-01 | 47.70 | 9.10 | 0.67 | 1.23 | 0.67 | 0.81 |
6.2 Daten filtern und Monatsdurchschnitt berechnen
# Gruppierung nach dem 'time'-Index und Berechnung des Durchschnitts für jede Gruppe
filtered_df_average = filtered_df.groupby(level='time').mean()
filtered_df_average = filtered_df_average.drop(columns=['lat', 'lon'])
# DataFrame anzeigen
df_wrapped = wrap_column_names(filtered_df_average, width=11)
df_wrapped.head()
| HWD_EU_clim ate_rcp45_m ean | HWD_EU_clim ate_rcp45_s tdev | HWD_EU_clim ate_rcp85_m ean | HWD_EU_clim ate_rcp85_s tdev | |
|---|---|---|---|---|
| time | ||||
| 1986-01-01 | 0.67 | 1.14 | 0.67 | 0.73 |
| 1987-01-01 | 0.79 | 1.14 | 0.79 | 0.73 |
| 1988-01-01 | 0.92 | 1.14 | 0.92 | 0.73 |
| 1989-01-01 | 0.93 | 1.14 | 0.93 | 0.73 |
| 1990-01-01 | 0.95 | 1.14 | 0.95 | 0.73 |
6.3 Definieren einer Funktion zur Erstellung eines Liniendiagramms mit Fehler
import matplotlib.pyplot as plt
import matplotlib.ticker as ticker
def plot_with_shaded_area(ax, x, y_mean, y_stdev, line_color, fill_color, line_label, fill_label, marker_style):
"""Hilfsfunktion zum Plotten von Mittelwertlinien mit schattiertem Fehlerbereich."""
ax.plot(x, y_mean, color=line_color, label=line_label, marker=marker_style, markevery=5, linestyle='--')
ax.fill_between(x, y_mean - y_stdev, y_mean + y_stdev, color=fill_color, alpha=0.3, label=fill_label)
def plot_line_and_shade(filtered_df_average, variable_name_list):
# Diagramm erstellen
fig, ax = plt.subplots(figsize=(13, 7), facecolor='#f1f1f1', edgecolor='k')
y_max_list = []
y_min_list = []
for variable_name in variable_name_list:
# Daten für RCP4.5 plotten
plot_with_shaded_area(
ax=ax,
x=filtered_df_average.index,
y_mean=filtered_df_average[f"{variable_name}_rcp45_mean"],
y_stdev=filtered_df_average[f"{variable_name}_rcp45_stdev"],
line_color='#1f77b4',
fill_color='#aec7e8',
line_label=f"{variable_name}_rcp45_mean",
fill_label=f"{variable_name}_rcp45_stdev",
marker_style=None
)
# Daten für RCP8.5 plotten
plot_with_shaded_area(
ax=ax,
x=filtered_df_average.index,
y_mean=filtered_df_average[f"{variable_name}_rcp85_mean"],
y_stdev=filtered_df_average[f"{variable_name}_rcp85_stdev"],
line_color='#ff7f0e',
fill_color='#ffbb78',
line_label=f"{variable_name}_rcp85_mean",
fill_label=f"{variable_name}_rcp85_stdev",
marker_style=None
)
# Dynamische Anpassung der Y-Achse
interval = 1
rcp45_min = filtered_df_average[f"{variable_name}_rcp45_mean"].min() - \
filtered_df_average[f"{variable_name}_rcp45_stdev"].max()
rcp85_min = filtered_df_average[f"{variable_name}_rcp85_mean"].min() - \
filtered_df_average[f"{variable_name}_rcp85_stdev"].max()
y_min = min(rcp45_min, rcp85_min) - 0.5
y_min = y_min // interval * interval
rcp45_max = filtered_df_average[f"{variable_name}_rcp45_mean"].max() + \
filtered_df_average[f"{variable_name}_rcp45_stdev"].max()
rcp85_max = filtered_df_average[f"{variable_name}_rcp85_mean"].max() + \
filtered_df_average[f"{variable_name}_rcp85_stdev"].max()
y_max = max(rcp45_max, rcp85_max) + 0.5
y_max = (y_max + interval) // interval * interval
y_max_list.append(y_max)
y_min_list.append(y_min)
ax.set_ylim(min(y_min_list), max(y_max_list))
# X-Achse für bessere Lesbarkeit anpassen
ax.set_xlim(filtered_df_average.index.min(), filtered_df_average.index.max())
ax.set_xticks(filtered_df_average.index[::5])
tick_positions = filtered_df_average.index[::5]
tick_labels = [str(pd.to_datetime(date).year) for date in tick_positions]
ax.set_xticks(ticks=tick_positions, labels=tick_labels, rotation=0)
# Gitterlinien hinzufügen
ax.grid(visible=True, color='#b0b0b0', linestyle='--', linewidth=0.8, alpha=0.6)
ax.yaxis.set_major_formatter(ticker.FormatStrFormatter('%0.2f'))
# Achsenbeschriftungen und Titel anpassen
ax.set_xlabel('Jahr', fontsize=14)
ax.set_ylabel('Temperatur (°C)', fontsize=14, labelpad=10)
ax.set_title(
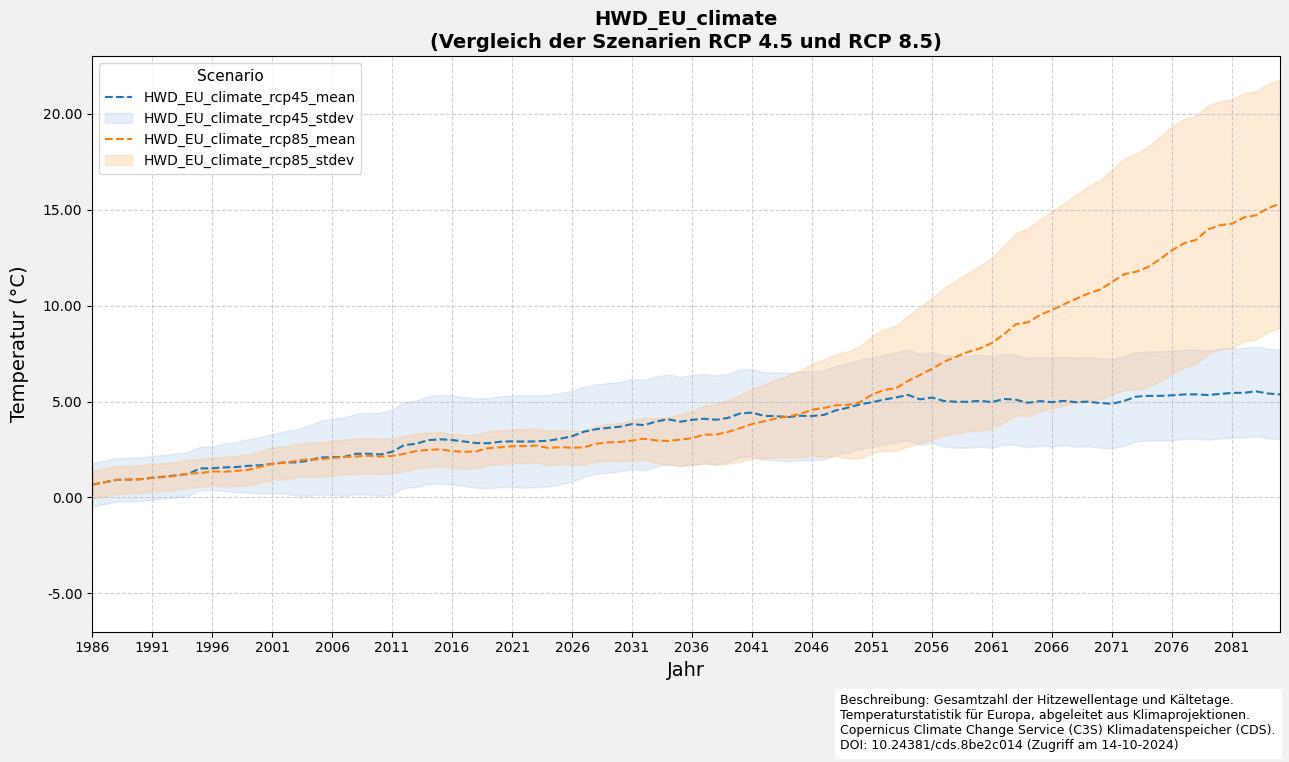
f"{variable_name}\n(Vergleich der Szenarien RCP 4.5 und RCP 8.5)",
fontsize=14,
fontweight='bold'
)
# Beschreibung und Quelle hinzufügen
plt.figtext(
0.65,
-0.04,
(
'Beschreibung: Gesamtzahl der Hitzewellentage und Kältetage.\n'
'Temperaturstatistik für Europa, abgeleitet aus Klimaprojektionen.\n'
'Copernicus Climate Change Service (C3S) Klimadatenspeicher (CDS).\n'
'DOI: 10.24381/cds.8be2c014 (Zugriff am 14-10-2024)'
),
ha='left',
va='center',
fontsize=9,
wrap=True,
backgroundcolor='w',
)
# Legende Anpassungen
ax.legend(loc='upper left', fontsize=10, frameon=True, title='Scenario', title_fontsize=11)
# Layout anpassen und Diagramm anzeigen
fig.tight_layout()
plt.show()
6.4 Visualisierung des Liniendiagramms mit Fehler
if __name__ == "__main__":
# Variable des Einzeldatensatzes
# Dieser Code schlägt für Cold Spell Days fehl, weil die Daten für Konstanz leer sind
plot_line_and_shade(filtered_df_average=filtered_df_average,
variable_name_list=[f"{nc_files[0]['variable_name']}"])
6.5 Definieren eine Funktion zur Erstellung einer Heatmap
import matplotlib.pyplot as plt
import cartopy.feature as cfeature
import cartopy.crs as ccrs
import numpy as np
def main_plt_plot(
nc_file=nc_file,
selected_year=None,
bounding_box=None):
# Öffnet die NetCDF-Datei
nc_dataset = nc.Dataset(nc_file['path'], mode='r')
lat = nc_dataset.variables['lat'][:]
lon = nc_dataset.variables['lon'][:]
# Falls eine Begrenzungsbox angegeben wurde, filtere die Daten entsprechend
if bounding_box:
lat_indices = np.where((lat >= bounding_box[1]) & (lat <= bounding_box[3]))[0]
lon_indices = np.where((lon >= bounding_box[0]) & (lon <= bounding_box[2]))[0]
lat_subset = lat[lat_indices]
lon_subset = lon[lon_indices]
else:
lat_indices = slice(None)
lon_indices = slice(None)
lat_subset = lat
lon_subset = lon
# Extrahiere Variablen-Daten
variable_name = nc_file['variable_name']
variable_data = nc_dataset.variables[variable_name][..., lat_indices, lon_indices]
var_units = getattr(nc_dataset.variables[variable_name], "units", "N/A")
# Bestimmen Sie den Jahresindex auf der Grundlage von selected_year.
if selected_year < 1986:
year_index = 0
year = 1985
elif selected_year > 2085:
year_index = -1
year = 2085
else:
year_index = selected_year-1986
year = selected_year
# Extrahieren Sie die Daten für das ausgewählte Jahr.
band_data = variable_data[year_index]
# NaN-Werte für Perzentilberechnungen entfernen
band_data_nonan = band_data[~np.isnan(band_data)]
vmin = np.nanpercentile(band_data_nonan, 1)
vmax = np.nanpercentile(band_data_nonan, 99)
def dynamic_round(value):
# Bestimmen Sie die Größe des Wertes.
order_of_magnitude = np.floor(np.log10(abs(value)))
# Verwenden Sie diese Größe, um die Genauigkeit dynamisch zu wählen.
if order_of_magnitude < -2: # Werte kleiner als 0,01
return round(value, 3)
elif order_of_magnitude < -1: # Werte zwischen 0,01 und 1
return round(value, 2)
elif order_of_magnitude < 0: # Werte zwischen 1 und 10
return round(value, 1)
else: # Werte 10 oder größer
return round(value)
# Dynamische Rundung auf vmin und vmax anwenden
vmin = dynamic_round(vmin)
vmax = dynamic_round(vmax)
bins = 10
interval = (vmax - vmin) / bins
# Erstellen Sie ein 2D-Netzgitter für die grafische Darstellung.
lon_grid, lat_grid = np.meshgrid(lon_subset, lat_subset)
# Erstelle die Figur
fig, ax = plt.subplots(
figsize=(12, 8),
facecolor='#f1f1f1',
edgecolor='k',
subplot_kw={'projection': ccrs.PlateCarree()}
)
# Kartenmerkmale hinzufügen
ax.coastlines(edgecolor='black', linewidth=1.5)
ax.add_feature(cfeature.BORDERS, edgecolor='black', linewidth=1.5)
# Erstellen Sie ein Farbnetzdiagramm mit der angegebenen Farbkarte und den Grenzen.
cmap = plt.get_cmap("viridis", bins)
pcm = ax.pcolormesh(
lon_grid,
lat_grid,
band_data,
transform=ccrs.PlateCarree(),
cmap=cmap,
shading='auto',
vmin=vmin,
vmax=vmax,
)
# Einen Farbbalken hinzufügen
ticks = np.linspace(vmin, vmax, num=bins + 1)
cbar = plt.colorbar(pcm, ax=ax, orientation='vertical', pad=0.02, ticks=ticks)
cbar.set_label(f"{variable_name}", fontsize=12)
cbar.ax.tick_params(labelsize=12)
# Gitterlinien hinzufügen
gl = ax.gridlines(draw_labels=True,
crs=ccrs.PlateCarree(),
linewidth=0.8,
color='gray',
alpha=0.7,
linestyle='--')
gl.top_labels = False
gl.right_labels = False
gl.xlabel_style = {'size': 10, 'color': 'black'}
gl.ylabel_style = {'size': 10, 'color': 'black'}
# Titel und Beschriftungen hinzufügen
fig.text(0.5, 0.0, 'Longitude', ha='center', fontsize=14)
fig.text(0.06, 0.5, 'Latitude', va='center', rotation='vertical', fontsize=14)
ax.set_aspect("equal")
# Einen Titel hinzufügen
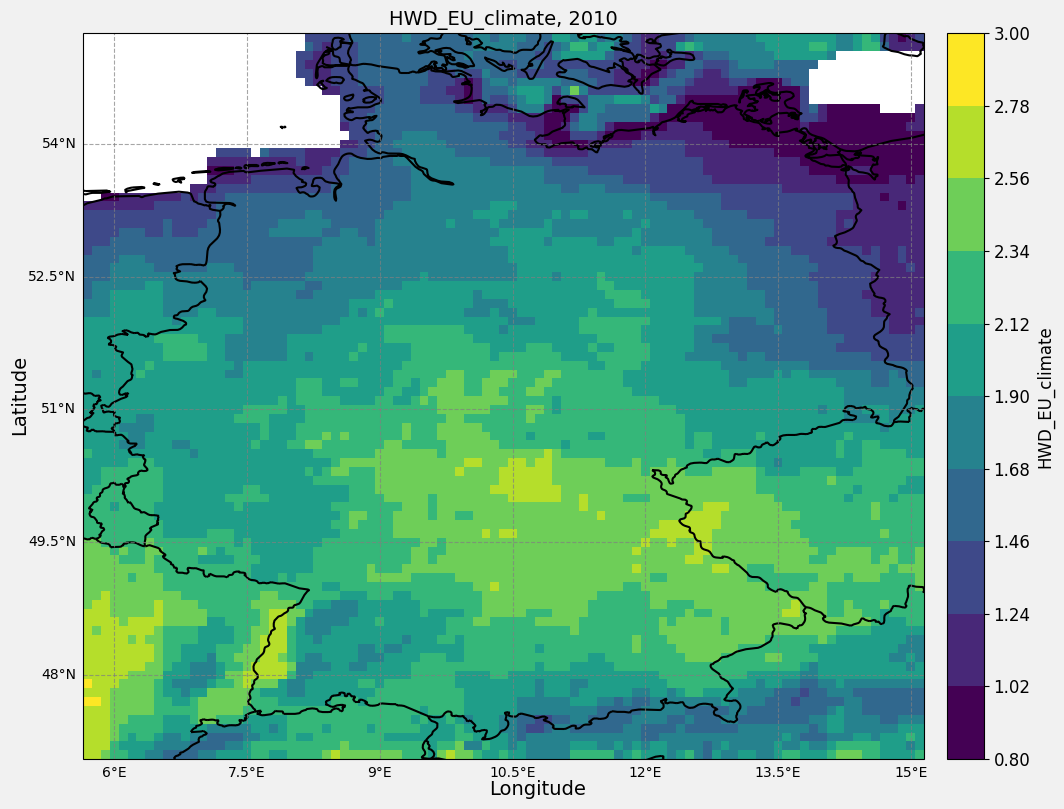
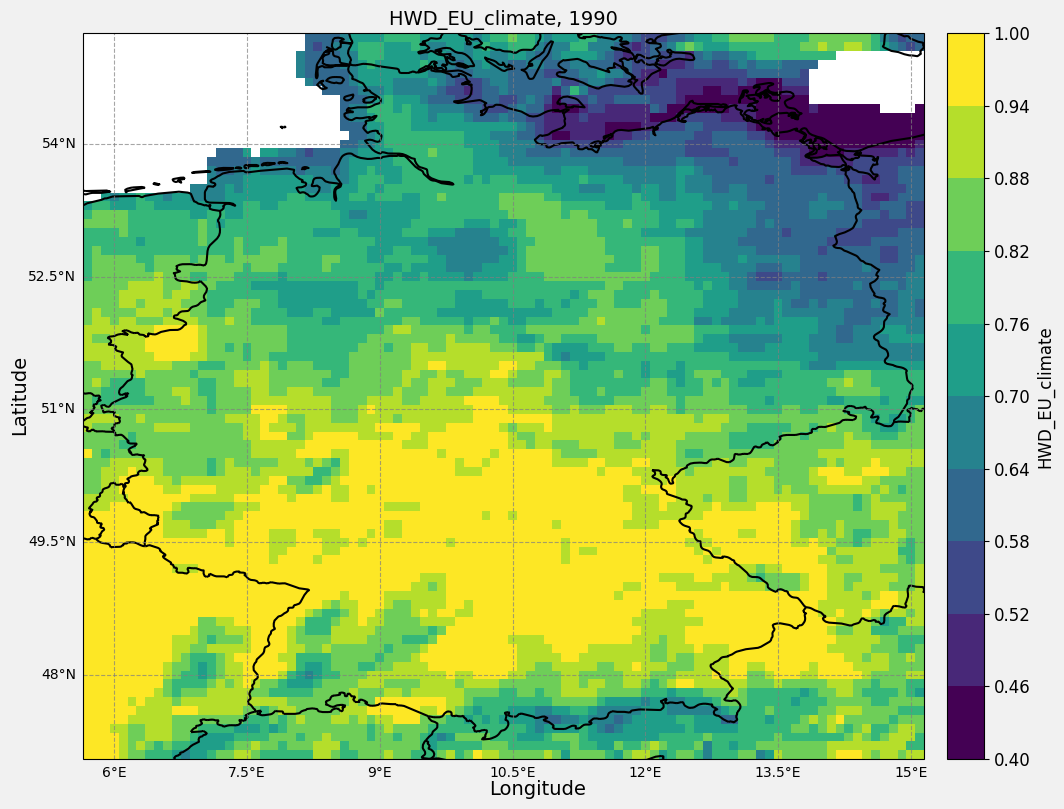
ax.set_title(f"{variable_name}, {year}", fontsize=14)
# Layout anpassen und das Diagramm anzeigen
plt.tight_layout()
plt.show()
6.6 Visualisierung mit Heatmap
if __name__ == "__main__":
# Beispiel für einen Anwendungsfall
main_plt_plot(nc_file=nc_files[0],
selected_year=1990,
)
main_plt_plot(nc_file=nc_files[0],
selected_year=2010,
)
F:\ProgramFiles\condaEnvs\cds_env\lib\site-packages\numpy\lib\_function_base_impl.py:4842: UserWarning: Warning: 'partition' will ignore the 'mask' of the MaskedArray.
arr.partition(
F:\ProgramFiles\condaEnvs\cds_env\lib\site-packages\numpy\lib\_function_base_impl.py:4842: UserWarning: Warning: 'partition' will ignore the 'mask' of the MaskedArray.
arr.partition(